Anndata(AnnData)是一个Python库,用于处理和分析单细胞RNA测序数据以及其他高维生物数据的工具。Anndata 是 scanpy(单细胞RNA测序分析工具)的基础数据结构,提供了一种方便的方式来存储、管理和操作单细胞数据。以下是关于 Anndata 的详细介绍:
数据结构
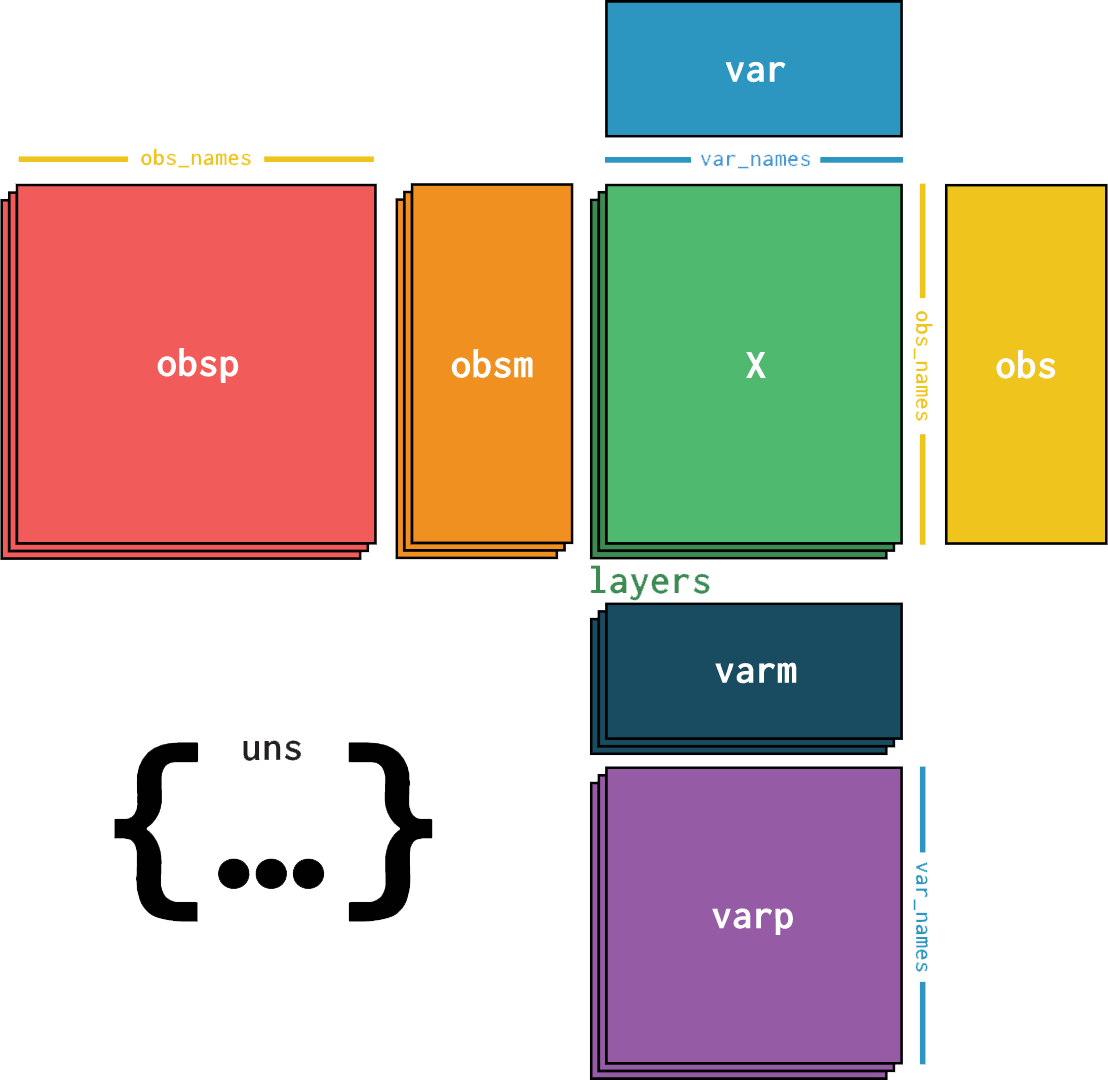
Anndata 是一个表格式的数据结构,类似于数据帧(DataFrame),但专门用于高维生物数据。它包括以下主要组件:
- X(数据矩阵): .X这个部分储存的是矩阵信息,数据结构是numpy array,和seurat对象一样,基因variable *细胞observation的稀疏矩阵。
但是.X的结构ndarray,是一个数组,是没有行名列名消息的。行名和列名消息存储在.obs和.var里。
obs(观测信息): .obs存储的是细胞的信息,数据结构是pandas dataframe。
相当于Seurat对象中的metadata。obsm:对观测的多维的注释,m指的就是multi-dim多个维度。它是2-多维的ndarray,长度为n_obs。(obs是一个维度可以都放在一个数据框下)
var(变量信息): .var存储的是基因的信息,数据结构是pandas dataframe。
layers(层): 可以存储其他数据层,如归一化后的数据或差异表达分析的结果。在做速率分析的时候,还可以看到adata中有layers这一部分的信息。
uns(未排序的数据): .uns存储的是后续添加的非结构信息,数据结构是dict,有序字典。
操作
创建 Anndata 对象:
import anndata as ad
import numpy as np
# 创建一些数据
data = np.random.randn(100, 4) # 100 个细胞和 4 个基因
obs = {'cell_type': ['type1', 'type2', 'type1', 'type2']*25} # 每个细胞的类型
var = {'gene_id': ['gene1', 'gene2', 'gene3', 'gene4']} # 每个基因的ID
# 创建 AnnData 对象
adata = ad.AnnData(X=data, obs=obs, var=var)
# 查看 AnnData 对象
print(adata)
adata
# AnnData object with n_obs × n_vars = 2638 × 1838
# obs: 'n_genes', 'n_genes_by_counts', 'total_counts', 'total_counts_mt', 'pct_counts_mt', 'leiden', 'louvain', 'louvain_anno'
# var: 'gene_ids', 'n_cells', 'mt', 'n_cells_by_counts', 'mean_counts', 'pct_dropout_by_counts', 'total_counts', 'highly_variable', 'means', 'dispersions', 'dispersions_norm', 'mean', 'std'
# uns: 'hvg', 'leiden', 'leiden_colors', 'neighbors', 'pca', 'rank_genes_groups', 'umap', 'draw_graph', 'diffmap_evals', 'louvain', 'paga', 'louvain_sizes', 'louvain_colors', 'leiden_sizes'
# obsm: 'X_pca', 'X_umap', 'X_draw_graph_fa', 'X_diffmap'
# varm: 'PCs'
# obsp: 'connectivities', 'distances'
数据操作:Anndata 允许您执行多种数据操作,包括切片、过滤、转置、连接数据、添加元信息等。
# 切片数据
subset_data = adata[:, list_of_genes]
# 过滤细胞
adata = adata[adata.obs['quality'] > 0.9]
# 转置数据
adata_T = adata.T
数据可视化: Anndata 可以与 scanpy 或其他可视化工具结合使用,以可视化数据、绘制UMAP、t-SNE图等。
import scanpy as sc
sc.tl.pca(adata)
sc.pl.umap(adata, color='cell_type')
数据存储: Anndata 可以将数据存储为HDF5文件,以便将数据持久化和共享。
adata.write('my_data.h5ad')
其他操作
添加元数据: 可以通过修改 adata.obs 和 adata.var 来添加或更新关于细胞和基因的信息。
# 添加新的细胞信息
adata.obs['treatment'] = ['drug', 'control'] * 50
# 添加新的基因信息
adata.var['chromosome'] = ['chr1', 'chr1', 'chr2', 'chr2']
索引和选择数据: 可以使用类似 Pandas 的方式来选择数据
# 选择特定类型的细胞
adata_subset = adata[adata.obs['cell_type'] == 'type1']
# 选择特定的基因
adata_subset_genes = adata[:, adata.var['gene_id'].isin(['gene1', 'gene3'])]
保存和加载 Anndata 对象
# 保存到 .h5ad 格式
adata.write('my_dataset.h5ad')
# 加载数据
adata_loaded = ad.read_h5ad('my_dataset.h5ad')

